1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
|
### Focus sur l'europe Top 7
e <- dbSendQuery(c, "WITH d AS (SELECT date(dateRep) AS dateRep
, countriesAndTerritories AS pays
, SUM(cases) OVER ( PARTITION BY countriesAndTerritories
ORDER BY date(dateRep)) AS cum_c
, SUM(deaths) OVER ( PARTITION BY countriesAndTerritories
ORDER BY date(dateRep)) AS cum_d
FROM data
WHERE continentExp = 'Europe'
)
SELECT *
FROM d
WHERE pays IN (
SELECT pays
FROM (
SELECT pays
, MAX(cum_d)AS n
FROM d
GROUP BY pays
ORDER BY n DESC
LIMIT 7
)
)
")
e1 <- dbFetch(e)
dbClearResult(e)
e1$dateRep <- as.POSIXct(e1$dateRep)
ge1 <- ggplot(e1) +
aes(x = dateRep, y = cum_d, color = pays ) +
geom_line() +
labs(
x = NULL,
y = NULL,
title = "Nombre de morts"
)
###
e <- dbSendQuery(c, "WITH d AS (SELECT date(dateRep) AS dateRep
, countriesAndTerritories AS pays
, SUM(cases) OVER ( PARTITION BY countriesAndTerritories
ORDER BY date(dateRep)) AS cum_c
, SUM(deaths) OVER ( PARTITION BY countriesAndTerritories
ORDER BY date(dateRep)) AS cum_d
FROM data
WHERE continentExp = 'Europe'
)
SELECT *
FROM d
WHERE pays IN (
SELECT pays
FROM (
SELECT pays
, MAX(cum_c)AS n
FROM d
GROUP BY pays
ORDER BY n DESC
LIMIT 7
)
)
")
e2 <- dbFetch(e)
dbClearResult(e)
e2$dateRep <- as.POSIXct(e2$dateRep)
ge2 <- ggplot(e2) +
aes(x = dateRep, y = cum_c, color = pays ) +
geom_line() +
labs(
x = NULL,
y = NULL,
title = "Nombre de cas"
)
e12 <- grid.arrange(ge1, ge2, ncol = 1, nrow = 2) |












 Répondre avec citation
Répondre avec citation
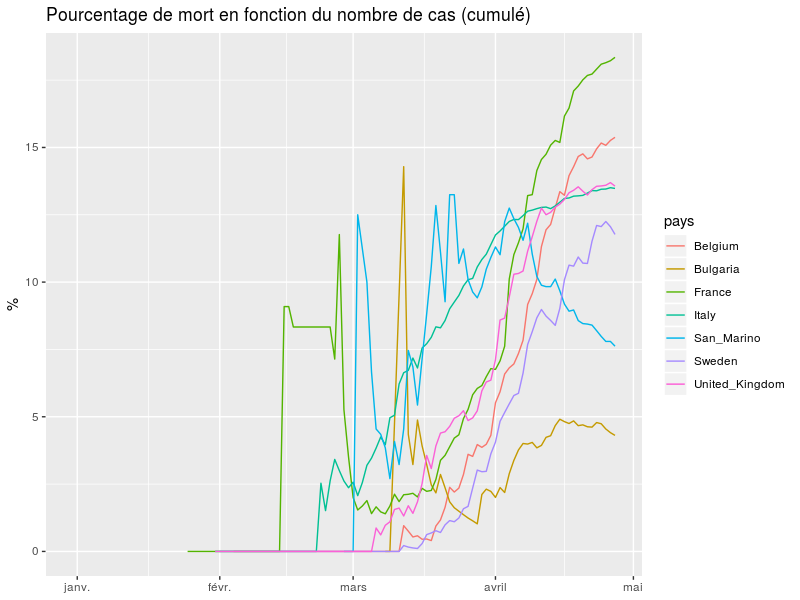
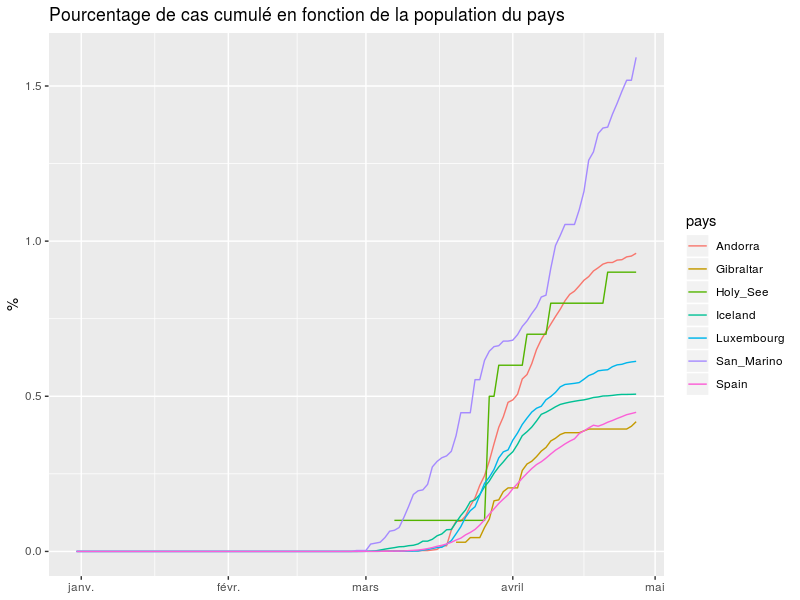
 Ce qui suis n'a pas l'objectif de prédire ni de mener a une quelconque conclusion juste a me distraire de mon insomnie. Et pourquoi pas d'inviter à faire vos propres visualisations de donnée, et éventuellement les partager.
Ce qui suis n'a pas l'objectif de prédire ni de mener a une quelconque conclusion juste a me distraire de mon insomnie. Et pourquoi pas d'inviter à faire vos propres visualisations de donnée, et éventuellement les partager.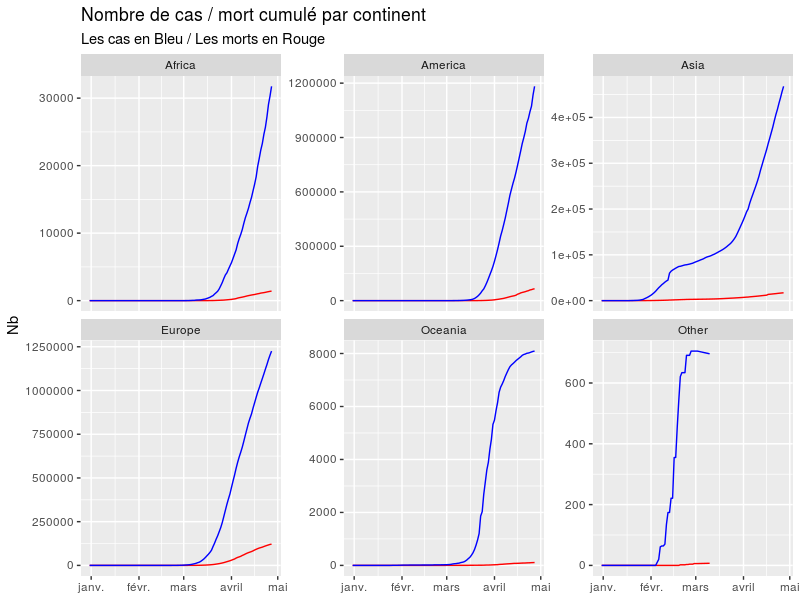
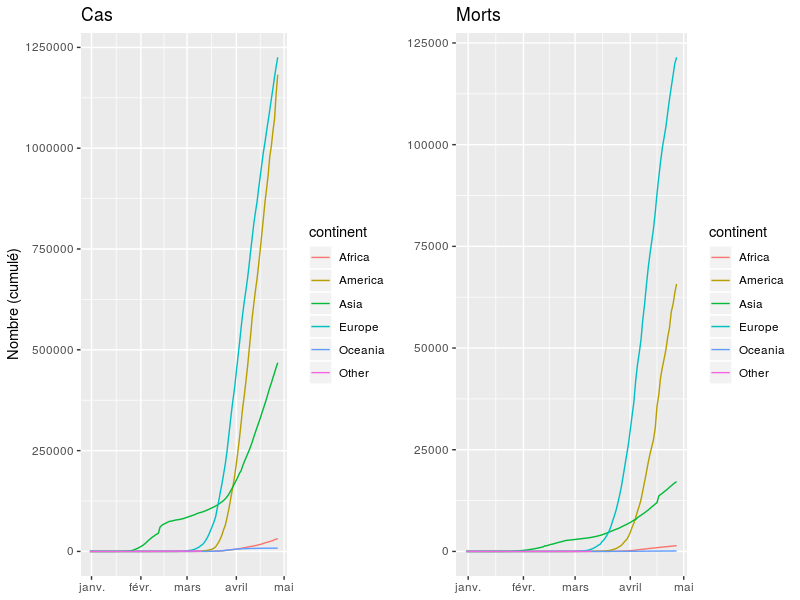
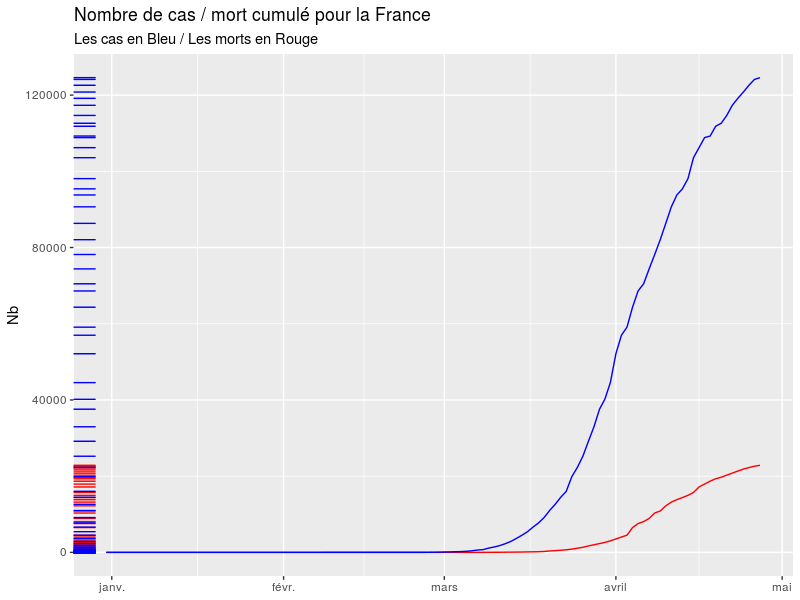
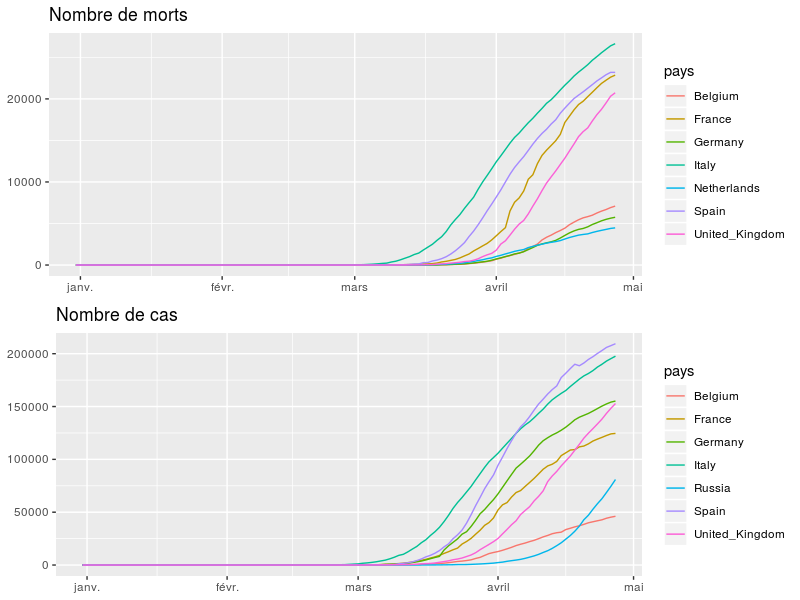

Partager