Bonjour,
Débutante avec R...
Je réalise un boxplot sur un dataset de données mais je souhaiterai filtrer les données et réaliser plusieurs boxplot pour séparer les données (je ne sais pas comment m'y prendre).
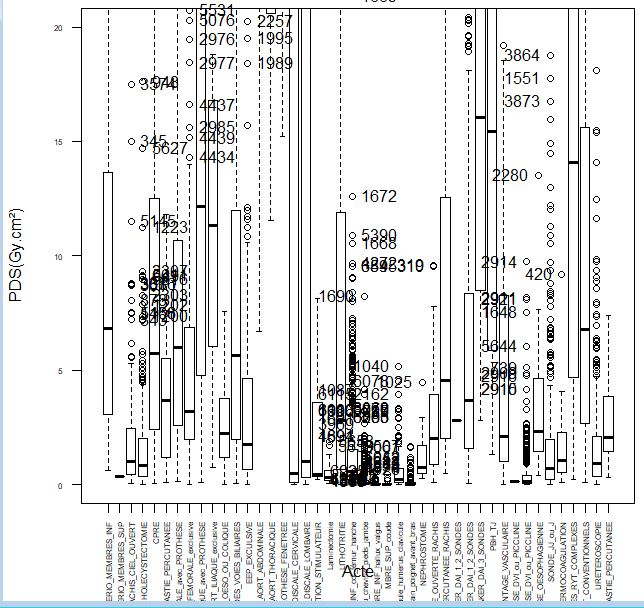
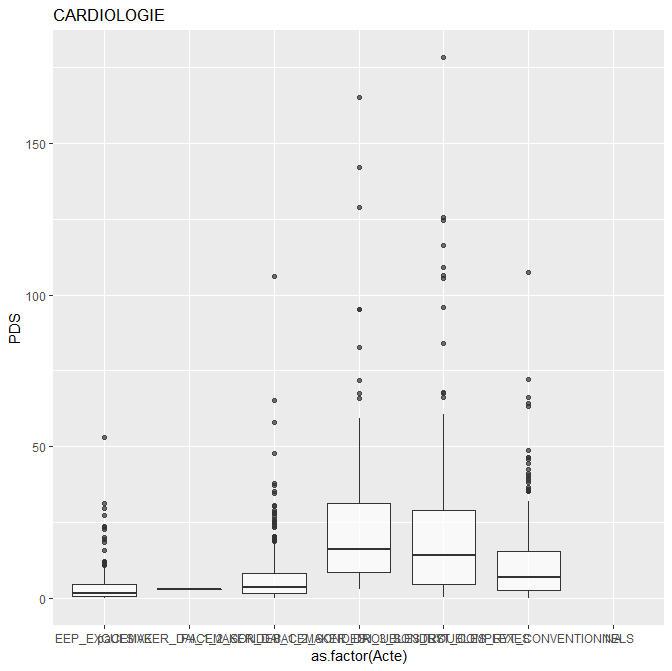
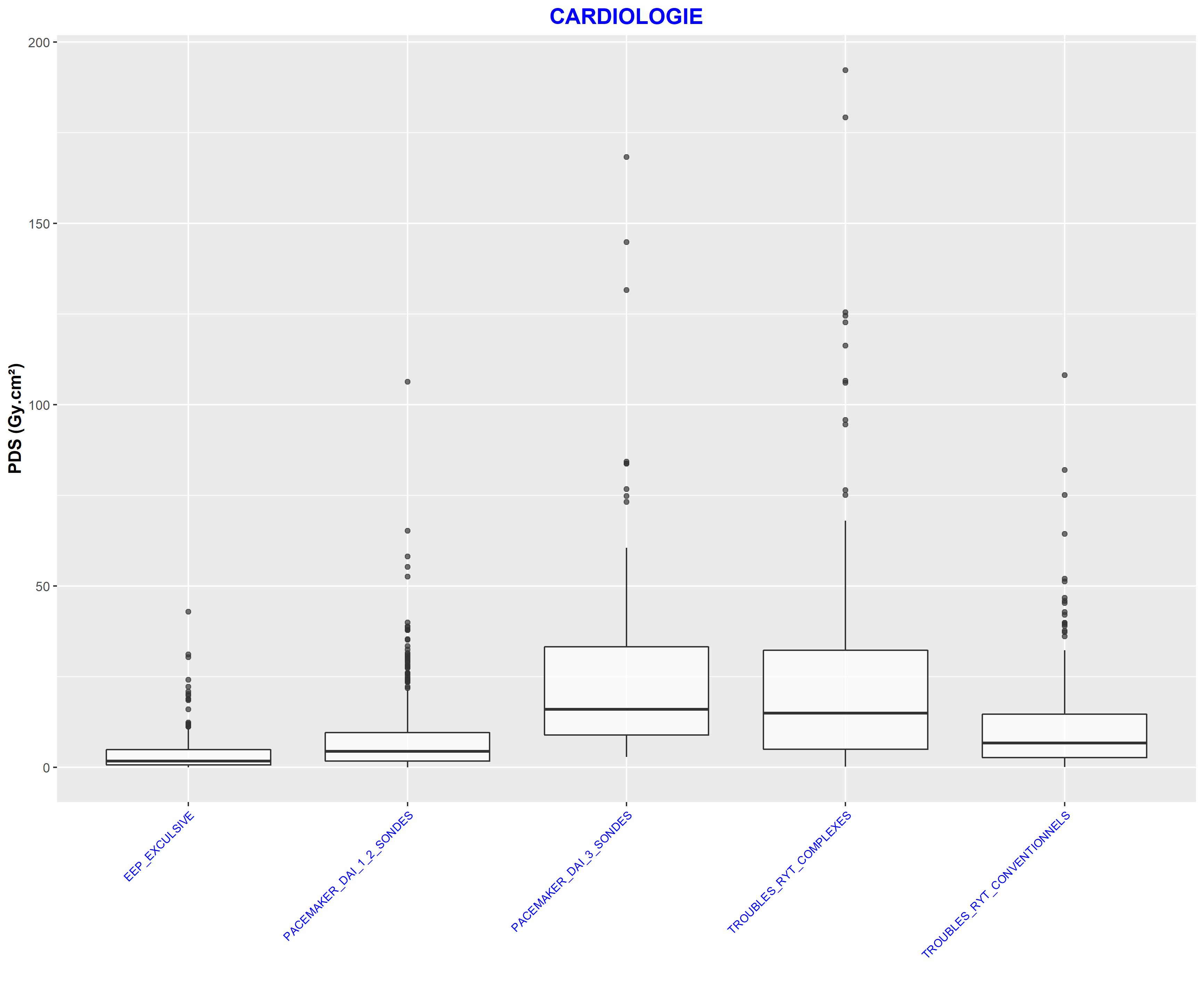
Le boxplot est réalisé sur la donnée "PDS" qui est la dose de RX délivrée pour un "acte" chirurgical.
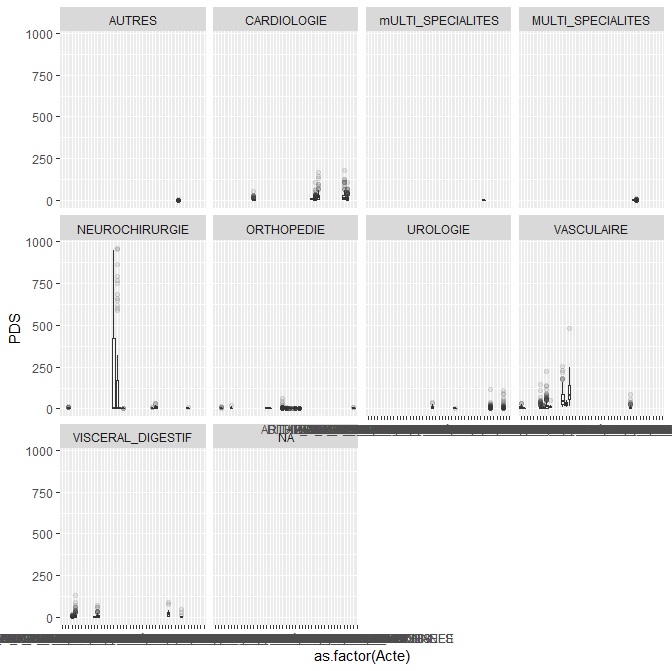
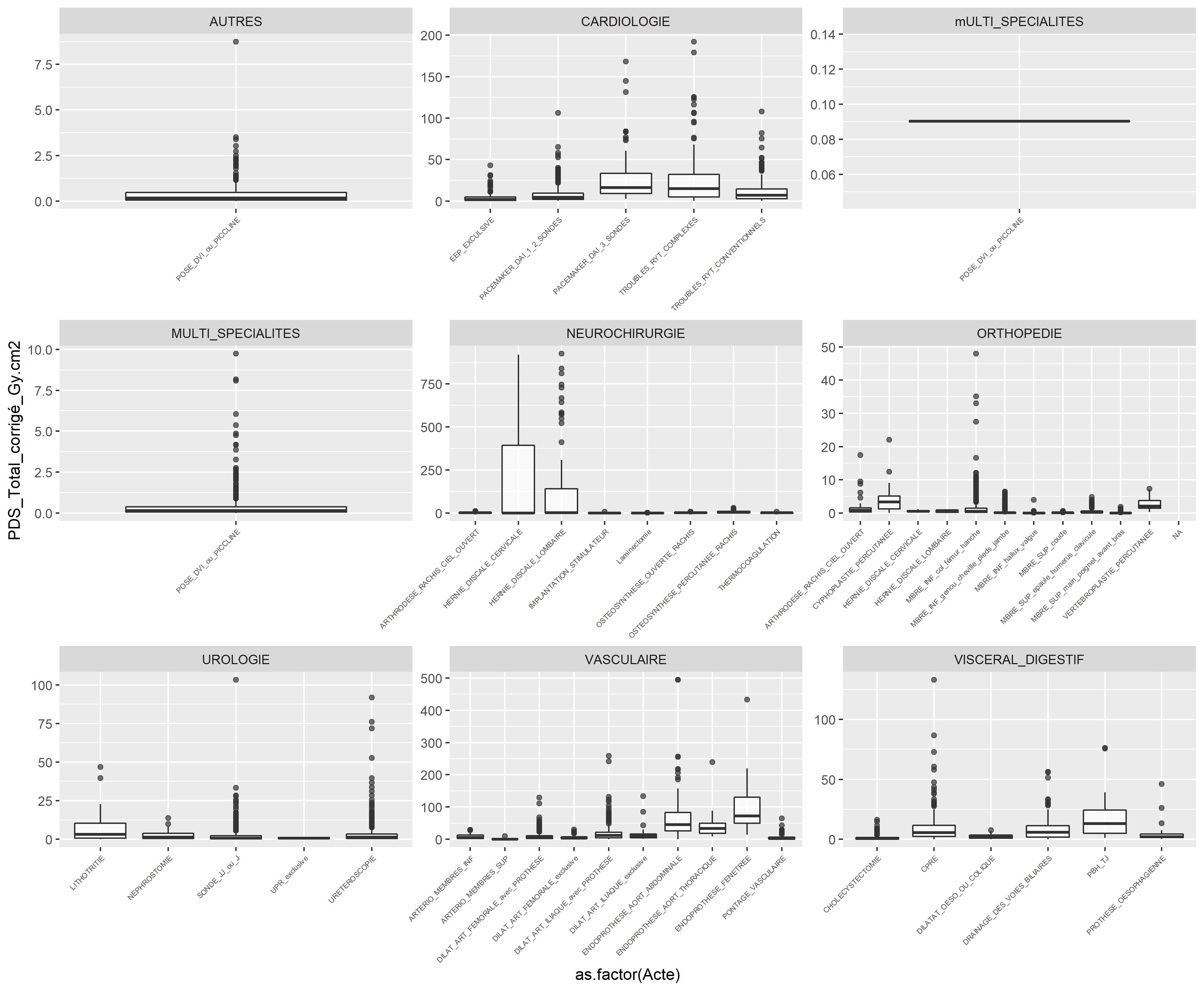
Dans mon tableau j'ai également une autre colonne qui spécifie la spécialité "Spe" chirurgicale et je voudrais filtrer par les données de cette colonne.
Comment faire ?
Voici les données de mon Dataset :
'data.frame': 8156 obs. :
….
$ Spe : Factor w/ 9 levels "AUTRES","CARDIOLOGIE",..: 9 9 9 9 9 9 9 9 9 9 ...
$ Acte : Factor w/ 44 levels "ARTERIO_MEMBRES_INF",..: 5 5 5 5 5 5 5 5 5 5 ...
$ PDS : num 1.7 2.77 2.7 5.18 7.82 ...
…..
Et voici comment je réalise mon Boxplot pour le moment :
Boxplot(PDS~Acte, data=Dataset, id=list(method="y"),ylim=c(0,20),las=2,cex.axis=0.5, ylab="PDS(Gy.cm²)")
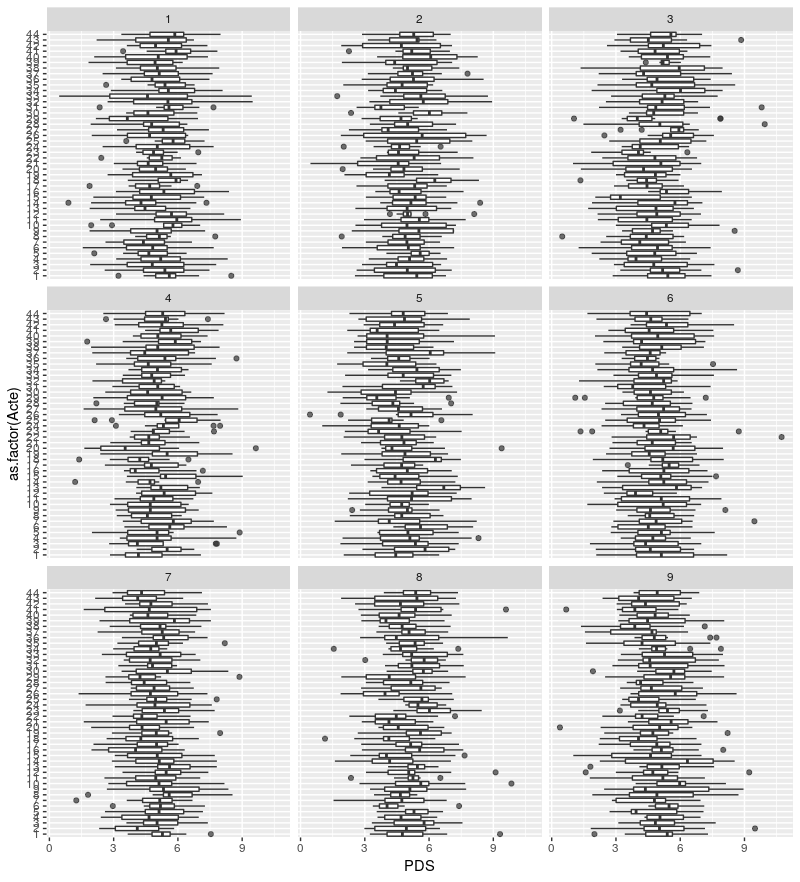
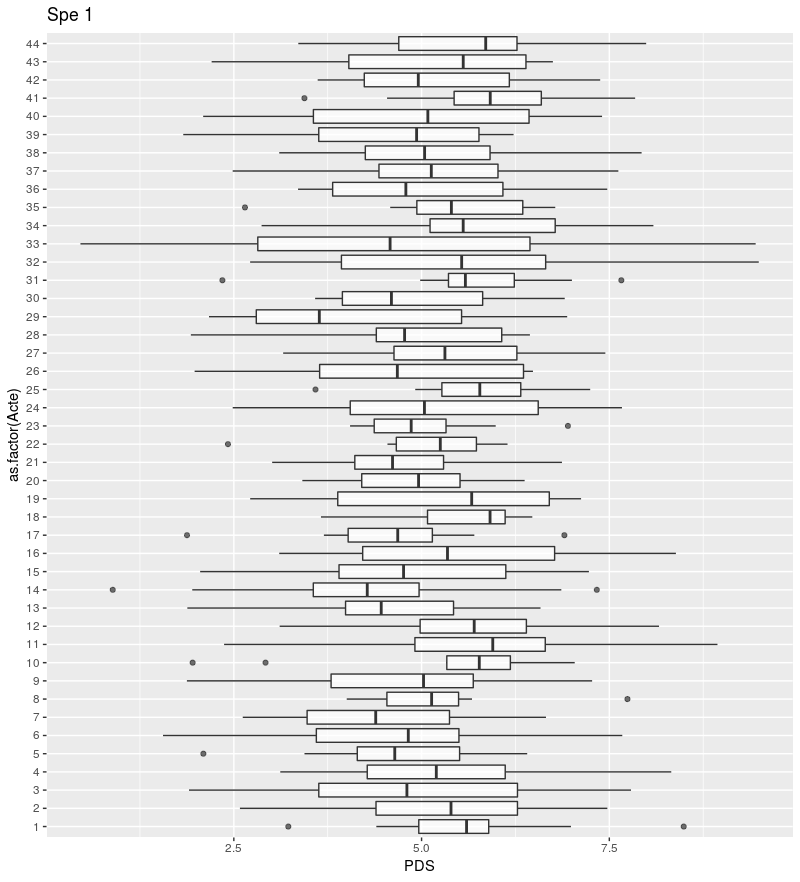
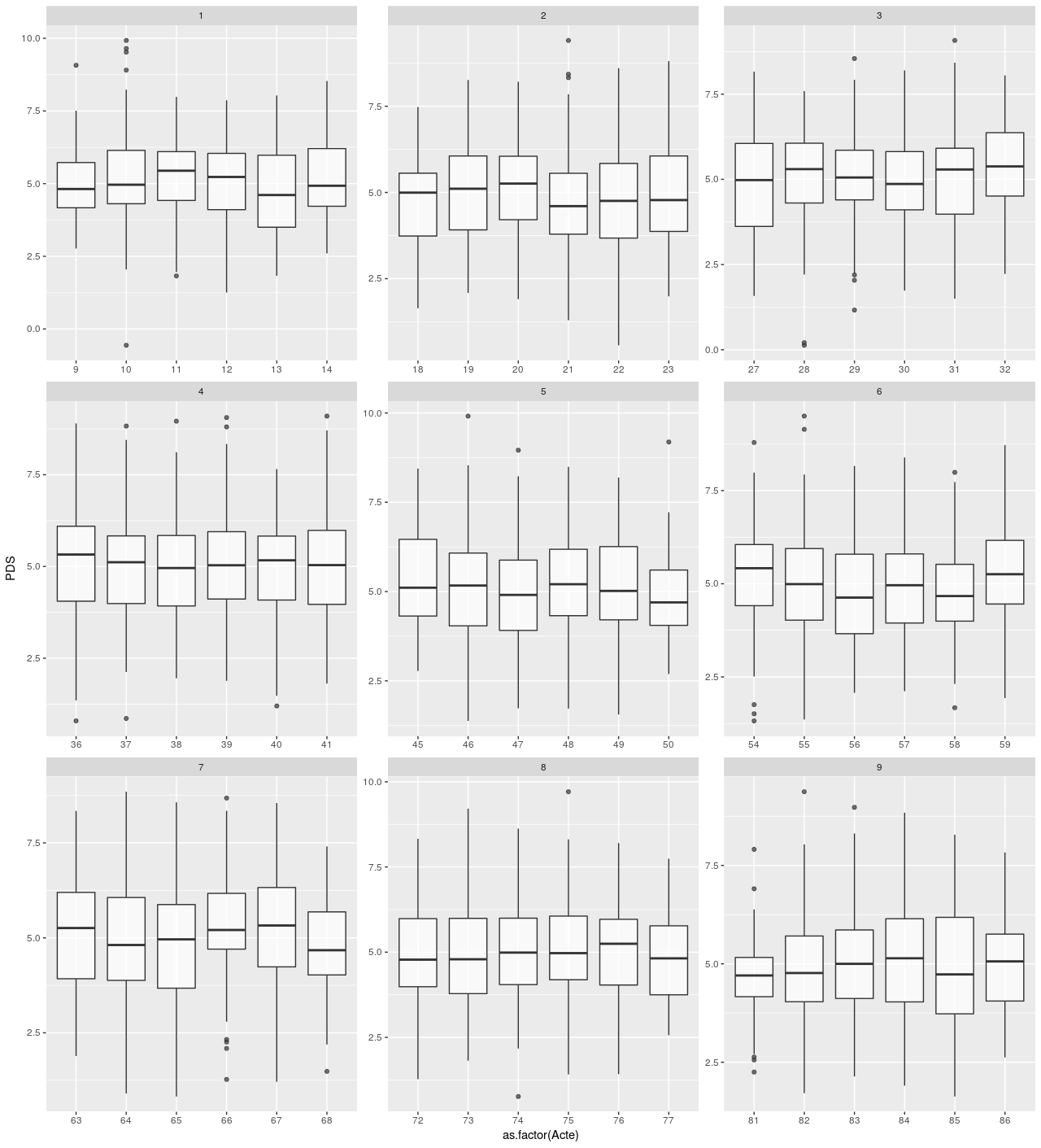
Mais j'ai besoin de faire plusieurs boxplot selon la colonne "Spe" qui regroupent 8 spécialités au bloc opératoire.
Je pensais essayer de filtrer pour faire mes 8 spécialités une par une avec un truc genre :
Boxplot(PDS~Acte + filter(Spe=="ORTHOPEDIE"), data=Dataset, id=list(method="y"),ylim=c(0,20),las=2,cex.axis=0.5, ylab="PDS(Gy.cm²)")
Mais cela ne marche pas….
Le top serait de faire 8 boxplot directement avec mes 8 spécialités chirurgicales…
Y a t-il un moyen ou une piste ?
Merci
Matou06
Voici mon boxplot :










 Répondre avec citation
Répondre avec citation



Partager