1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
|
invisible(sapply(c("drc","foreach", "magrittr"),
function(z) library(z, character.only = TRUE)))
ourData <- list(
x = c(1, 2, 2, 3, 4, 4, 4, 5, 5, 6, 6, 6, 6, 6, 7, 7, 7, 8, 8, 8, 8, 9, 10,
10, 11, 11, 12, 12, 12, 12),
y = c(4.9995, 4.9962, 4.9962, 4.9899, 4.9812, 4.9812, 4.9812, 4.9709, 4.9709,
4.9593, 4.9593, 4.9593, 4.9593, 4.9593, 4.9469, 4.9469, 4.9469, 4.9339,
4.9339, 4.9339, 4.9339, 4.9204, 4.9067, 4.9067, 4.8927, 4.8927, 4.8787,
4.8787, 4.8787, 4.8787)
)
visFitResult <- function(fitObj = NULL) {
oldPar <- par(las = 1)
if (is.null(fitObj)) {
plot(y ~ x, data = ourData, pch = 19L, col = "red")
} else {
plot(y ~ x, data = ourData, pch = 19L, col = "red")
plot(fitObj, add = TRUE, broken = TRUE, lty = 2L, lwd = 1, col = "red")
}
grid(20L, col = "gray")
par(oldPar)
invisible(TRUE)
}
visFitResult()
#drc::drm
drcObj <- drm(
formula = y ~ x,
data = ourData,
fct = weibull1(names = c("b=-d", "c=a", "d=a-b", "e=exp(-log(c) / b)"))
)
summary(drcObj)
visFitResult(drcObj)
#stats:nls: Self Start.
nlsObj <- list(
resWei = nls(
formula = y ~ SSweibull(x, Asym, Drop, lrc, pwr),
data = ourData,
control = nls.control(printEval = TRUE)
),
resBiexp = nls(
formula = y ~ SSbiexp(x, A1, lrc1, A2, lrc2),
data = ourData,
control = nls.control(printEval = TRUE)
),
resFpl = nls(
formula = y ~ SSfpl(x, A, B, xmid, scal),
data = ourData,
control = nls.control(printEval = TRUE)
),
resLog = nls(
formula = y ~ SSlogis(x, Asym, xmid, scal),
data = ourData,
control = nls.control(printEval = TRUE)
),
resMiMe = nls(
formula = y ~ SSmicmen(x, Vm, K),
data = ourData,
control = nls.control(printEval = TRUE)
)
)
summary(nlsObj$resWei)
nlsObj$resWei$m$getPars()
visFitResult(nlsObj$resWei)
#Erreurs. Celle de drc vient de summary(drcObj) mais on peut aussi la calculer
#manuellement en se servant des résultats obtenus
sapply(nlsObj, function(x) x$convInfo$finTol) %>% c(drc = 3.010583e-4) %>% sort
#Comportements des modèles obtenus avec des résultats pseudo-aléatoires
randomize <- function(size = 30L) {
oldPar <- par(las = 1L, mfcol = c(2L, 3L))
arg <-rlnorm(size)
plot(arg, drcObj$curve[[1]](arg),
xlab = "drc", ylab = "", pch = 20L)
foreach(i = nlsObj,
j = c("Weibull", "Biexponential", "Logistic-3par", "Logistic-4par",
"Michaelis-Menten")) %do% {
plot(arg, i$m$predict(arg),
xlab = j, pch = 20L, ylab = "", new = TRUE)
}
par(oldPar)
invisible(TRUE)
}
randomize() |









 Répondre avec citation
Répondre avec citation





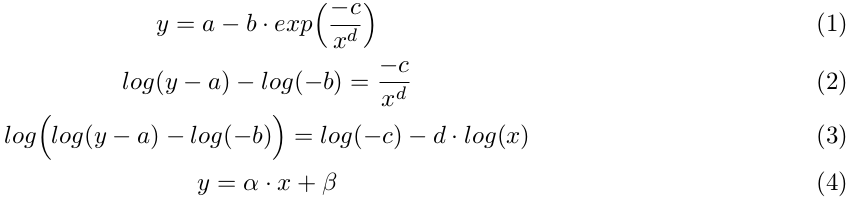

Partager