1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
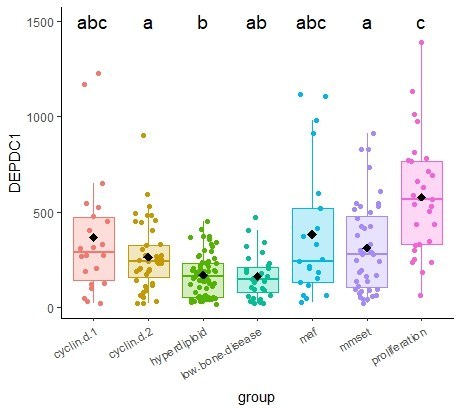
| pp <- pairwise.wilcox.test(myeloma$DEPDC1, myeloma$group,p.adjust.method ="holm" )
mymat <-tri.to.squ(pp$p.value)
library(multcompView)
# création des lettres correspondant à chaque moyenne
myletters <- multcompLetters(mymat,compare="<=",threshold=0.05,Letters=letters)
myletters
myletters_df <- data.frame(group=names(myletters$Letters),letter = myletters$Letters )
# plot
ggplot(myeloma, aes(x=group, y=DEPDC1, colour=group, fill=group))+
geom_boxplot(outlier.alpha = 0, alpha=0.25)+
geom_jitter(width=0.25)+
stat_summary(fun.y=mean, colour="black", geom="point",
shape=18, size=3) +
theme_classic()+
theme(legend.position="none")+
theme(axis.text.x = element_text(angle=30, hjust=1, vjust=1))+
geom_text(data = myletters_df, aes(label = letter, y = 1500 ), colour="black", size=5) |












 Répondre avec citation
Répondre avec citation





Partager