Bonjour, j'ai effectué un script en perl permettant de récupérer les régions spécifiques d'une souche bactérienne. Cependant je dois vérifier si les séquences sont vraiment spécifiques en faisant un blast de celle ci contre ma base de donnée.
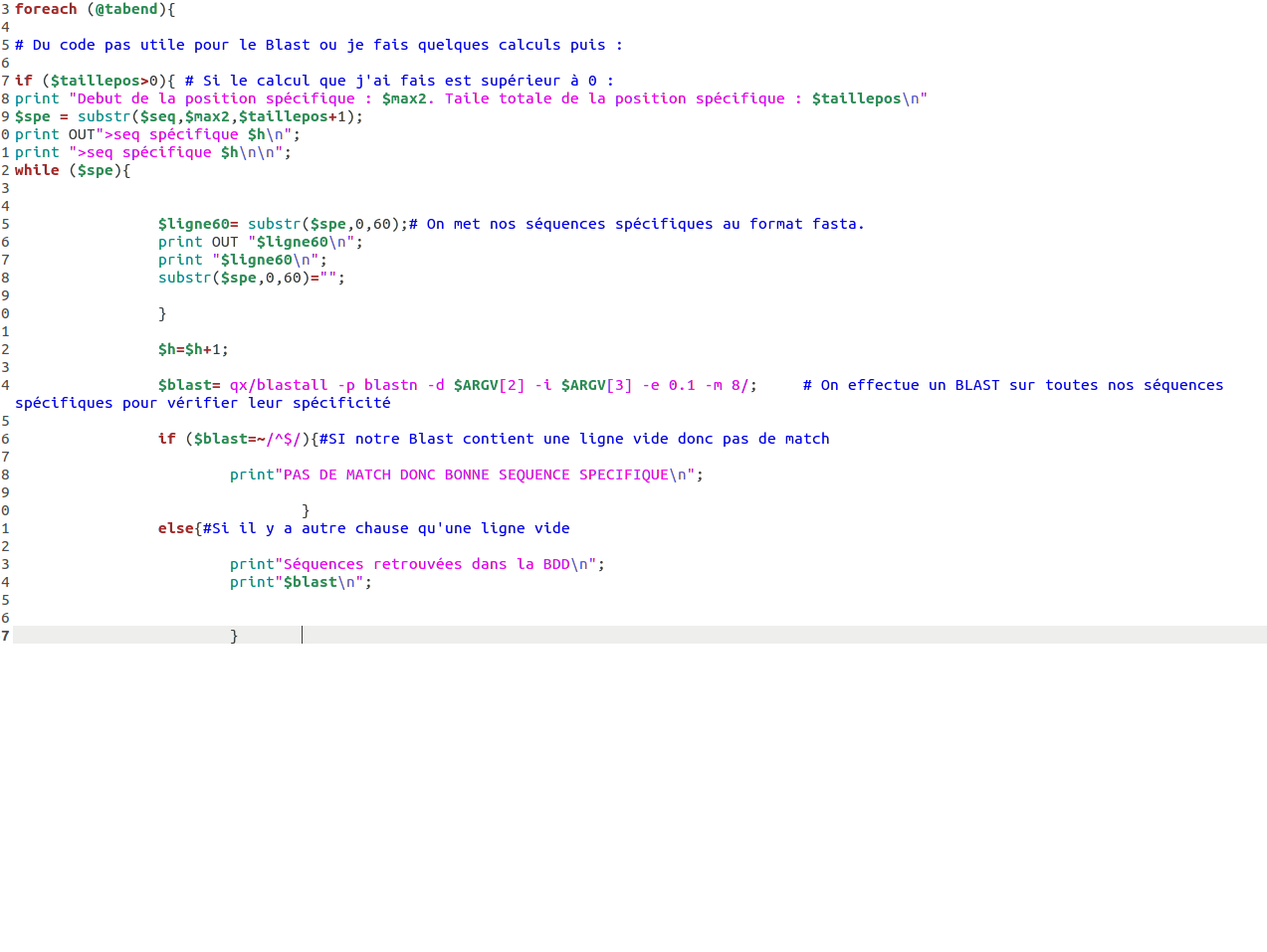
Mon problème et que lors de mon Blast une fois qu'il trouve une correspondance il l'applique jusqu'à la fin voici la partie Blast de mon code :
Pièce jointe 465818
Voici ce qu'il se passe sur mon fichier de sortie :
Pièce jointe 465815
On peut voir que dès qu'il y a une correspondance il l 'applique pour les résultats suivant . J'ai vérifier à la main pour la séquence spécifique 28 il n'y a normalement pas de correspondance.
Mon fichier £ARGV[3] contient toutes les séquences spécifique que j'ai récupéré au format fasta elles sont les unes à la suite des autres
Merci de votre réponse.









 Répondre avec citation
Répondre avec citation


Partager