Bonjour à vous,
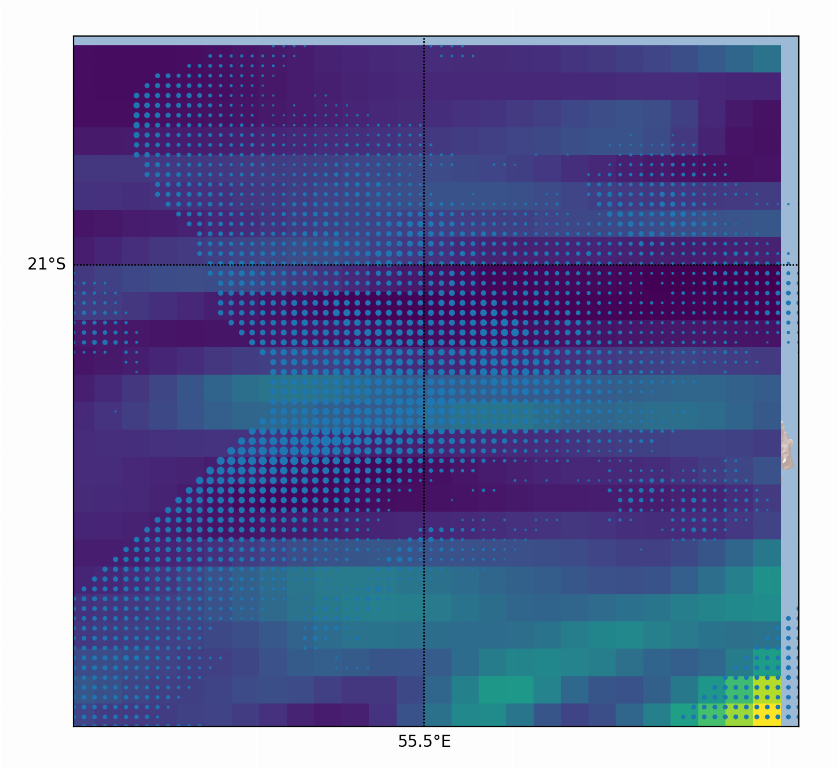
Je cherche à comparer deux champs (le premier est sous forme x y z , soit lon lat et valeur), et le deuxième champ est un champ 2D provenant d'un modèle numérique.
Les deux ont une zone en commun, mais les deux données ne sont pas de la même résolution, le plus résolu étant le premier (couvrant un champ 2D de 4950x4950 points) alors que le deuxième provenant du modèle numérique (1350 points sur 750).
Je suppose donc qu'il faut avoir une grille commune, idéalement sur le champ le moins résolu (du modèle numérique). Il faut donc réenchantillonner le premier champ.
Voici mon début de code, qui ne fonctionne pas en l'état
:
Premiere question : je n'arrive pas à mettre en 2D mon champ en x y z. Ensuite comment faire pour que ce champ corresponde à celui du modèle ? (arome ici)
Code : Sélectionner tout - Visualiser dans une fenêtre à part
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
Merci à vous pour vos pistes
Cordialement,
Jonathan






 Répondre avec citation
Répondre avec citation





Partager