Bonjour à tous,
dans le cadre de mes études je dois réaliser une segmentation automatique des tumeurs cérébrales dans une IRM.
lorsque j'applique un simple filtre sobel ou autre pour voir quelles sont les structures du cerveau détectés, j'ai remarqué qu'il n'arrive pas toujours à détecter les zones des tumeurs..
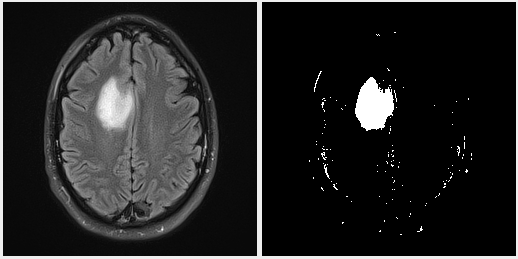
comme première solution j'ai essayé de réaliser une binarisation "manuelle"
exemple
ceci permet de mettre en valeur la zone de la tumeur pour mieux la détecter dans les autres traitements de mon code...
Code : Sélectionner tout - Visualiser dans une fenêtre à part
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
cependant cette fonction de "pretraitement" n'est pas automatique car pour chaque image je dois faire entrer les 2 valeurs V1 et V2....
je pense que je dois faire une étude statistique permettant de trouver une loi à laquelle je peux prévoir automatiquement pour chaque image les valeurs V1 et V2.. mais je ne sais pas par où je dois commencer et comment..
que pensez vous? et si vous avez d'autres propositions j'en suis preneuse
voilà image avant "pretraitement"
http://www.hostingpics.net/viewer.ph...96492avant.png
et image après "pretraitement"
http://www.hostingpics.net/viewer.ph...70425apres.png










 Répondre avec citation
Répondre avec citation













Partager