Bonsoir à toutes et à tous !!
Je dois faire des tests sur un dataset.
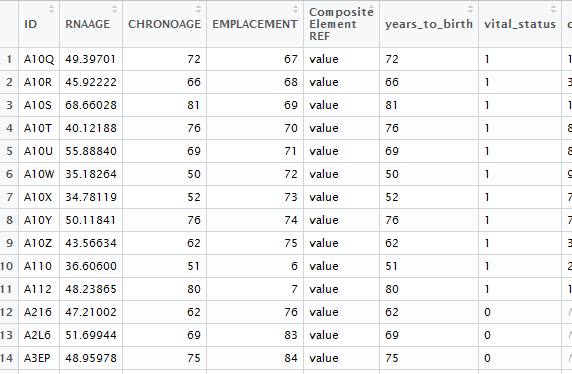
Voici une partie de mon dataset (appelé df_tumor) :
Je voudrais tester par exemple la normalité en utilisant shapiro.test de RNAage selon le statut vital (dont les valeurs sont 0 et 1), donc faire deux groupes, un groupe RNAAGE avec ceux qui ont une statut vital 0 et un groupe avec ceux qui ont un statut vital 1.
Je pensais faire quelque chose genre :
ouError in shapiro.test(RNAAGE ~ vital_status, df_tumor) :
unused argument (df_tumor)
Mais chaque fois j'ai une erreur, donc je me demandais si c'était possible ou pas !shapiro.test(df_tumor$RNAAGE ~ vital_status)
Error: is.numeric(x) n'est pas TRUE
Merci beaucoup pour votre aide !!!









 Répondre avec citation
Répondre avec citation

Partager