Bonjour à tous !
Je travaille sur scanners 3D ( j'ai à ma disposition pour chaque patient des CT de mise en place ou CTMEP, et les CBCT ). Je possède également des masques qui permettent d'afficher la ROI, la zone de la tumeur. Le but dans un premier temps était de superposer mon masque sur mon scanner afin d'avoir un scanner avec la région tumorale dessus.
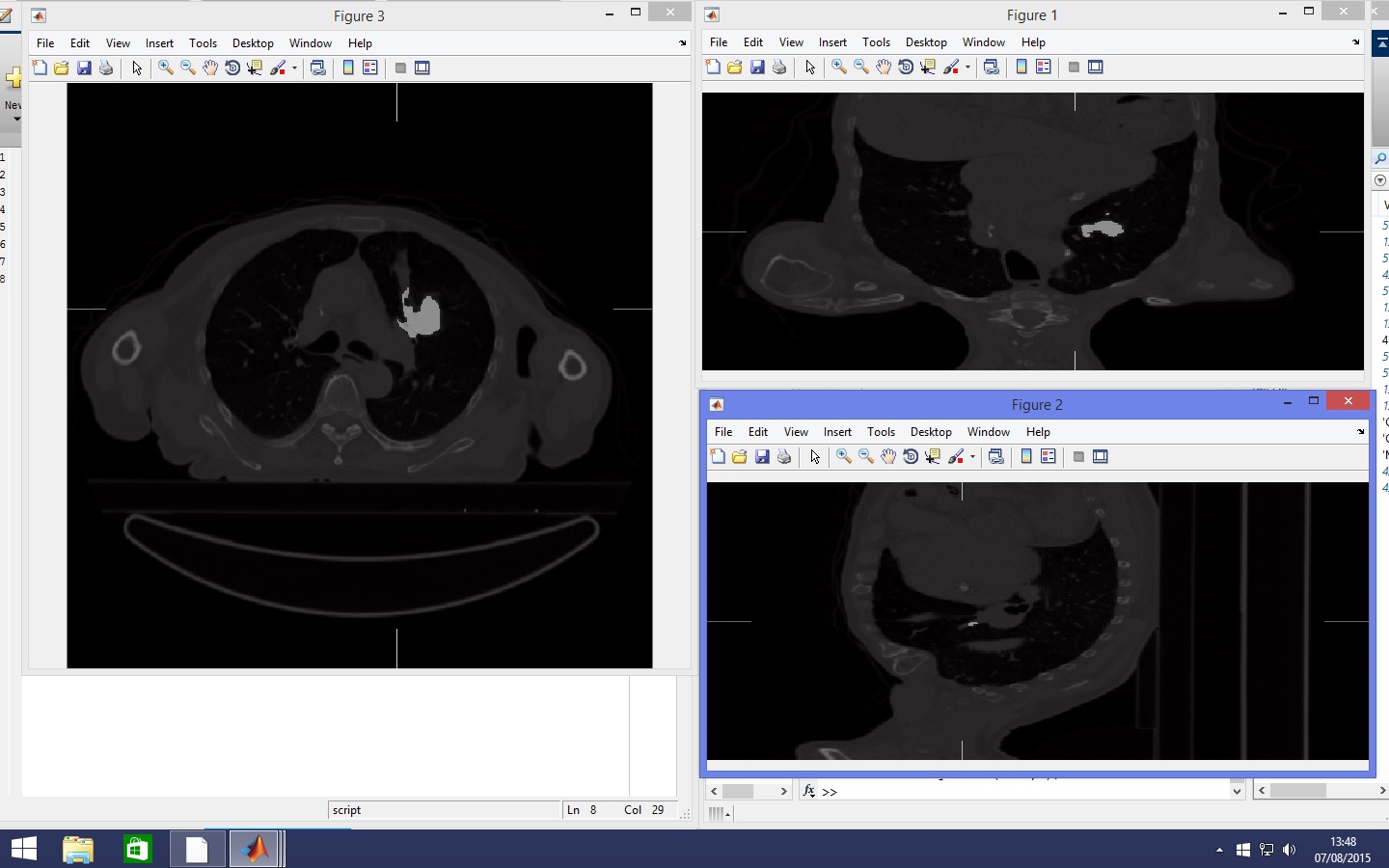
J'ai trouvé une fonction qui s'appelle showMaskAsOverlay, prennant deux images, et permet la superposition des deux. J'ai obtenu des résultats satisfaisants que vous pourrez vous sur ma première image " overlay" .
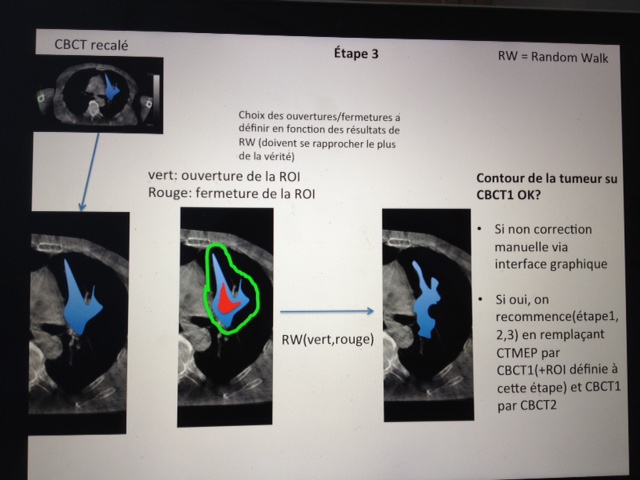
J'aimerais maintenant pour initialiser mes germes ( je travaille sur l'algorithme de marche aléatoire, prenant en entrée des " germes " à l'intérieur de ma ROI ,et à l'extérieur , comme vous pourrez le voir sur mon shéma " étape 3 " . L'érosion et la dilatation ( ou l'ouverture et la fermeture de la ROI ) vont me permettre de les initialiser. Le problème est que je ne vois pas comment m'en sortir. Il faut, je pense , que j'érode et je dilate mon masque de sorte à obtenir mes germes rouges et vertes mais je ne sais pas trop comment faire ....
J'aimerais de l'aide car je suis un peu perdu. Si vous avez besoin de plus d'explications sur certains points , je peux vous en donner.
Je vous mets également à disposition showMaskAsOverlay, au cas où vous voulez savoir comment j'ai obtenu ma superposition.
http://www.mathworks.com/matlabcentr...askAsOverlay.m
Merci d'avance..









 Répondre avec citation
Répondre avec citation
Partager